Über den Entwurf des Lebens: Mobile genetische Elemente (Experten)
Genetische Quellen der Anpassungsfähigkeit
Dieser Text stammt aus dem Studium Integrale Journal 30, https://www.si-journal.de/index2.php?artikel=jg30/heft1/sij301.html.
Peter Borger
Die DNA aller Organismen enthält eine große Anzahl genetischer Elemente, die heute als transponierbare und transponierte Elemente (TEs) bezeichnet werden. Einst als egoistische DNA interpretiert, stellen diese Elemente nach einer wachsenden Anzahl von Studien wichtige strukturelle und regulatorische Elemente des Genoms dar. Barbara McClintocks 80 Jahre alte Vorstellung von Transposons als „Kontrollelementen“ des Genoms hat sich zweifelsfrei bestätigt.

Introbild Dunkle und helle Varianten des Birkenspanners (Bistonbetularia). Die Unterschiede werden durch ein springendes Gen verursacht. (iredding01, AdobeStock)
Kompakt
In der biologischen Fachliteratur werden im Zusammenhang mit Anpassung und Artbildung zunehmend die Begriffe „fast“, „superfast“, „rapid“ und „speedy“ verwendet. Dies zeigt, dass Anpassung, aber auch Artbildung, auffällig schnell erfolgen. Das deutet darauf hin, dass programmierte Merkmale von Lebewesen ausgeprägt werden. Zum besseren Verständnis braucht es Klarheit darüber, wie Variation entsteht. Zufallsmutationen alleine sind nicht ausreichend. Die Erforschung und detaillierte Analyse von Tausenden von Genomen ermöglichten eine Vorstellung davon, wie solche superschnellen Prozesse möglich sind. Die DNA aller Organismen enthält eine große Anzahl genetischer Elemente, die heute als transponierbare und transponierte Elemente (TEs) bezeichnet werden. Die einfachsten sind bakterielle IS-Elemente (Insertionssequenzen, DNA-Transposons), die komplexesten sogenannte endogene Retroviren (ERV) und die „long interspersed nuclear elements“ (LINE). Einst als egoistische DNA interpretiert, stellen diese Elemente gemäß einer wachsenden Anzahl von Studien wichtige strukturelle und regulatorische Elemente des Genoms dar. Über Genverdopplungen und -verluste bestimmen sie die Größe und Struktur der Genome* und spielen eine entscheidende Rolle in der Steuerung der Gen-Expression*. Letzteres, weil diese Elemente mit Sequenzen gekoppelt sind, die als Bindungsstellen für Transkriptionsfaktoren fungieren. Das sind DNA-Sequenzen, die die Expression benachbarter Gene regulieren. Zwei Gruppen von Transposons können unterschieden werden: solche, die sich über ein DNA-Zwischenprodukt in einem Cut-Paste-Mechanismus bewegen (DNA-Transposons), und retrotransponierbare Elemente (Retrotransposons), die sich über einen Copy-Paste-Mechanismus bewegen, bei dem ein RNA-Zwischenprodukt beteiligt ist. Barbara McClintocks 80 Jahre alte Vorstellung von Transposons als „Kontrollelementen“ des Genoms hat sich zweifelsfrei bestätigt.
Variation und ihre Ursachen
Die Vielzahl von Varianten innerhalb von Arten veranlasste Darwin dazu, seine berühmte Evolutionshypothese aufzustellen. Obschon Darwin vermutete, dass Variation von natürlichen Gesetzen bestimmt würde, schrieb er 1859, dass es akzeptabel sei, Variation als eine Folge des Zufalls zu betrachten – ihre Ursache und Ursprung wären demnach unbekannt (Darwin 1859). Im Jahr 1871 schrieb er sogar, dass die „Geburt von Arten, wie auch die von Individuen … Teil derselben Aneinanderreihung von Ereignissen [ist], die leider nicht durch unser Gehirn als die Folge eines blinden Zufallsprozesses akzeptiert werden will“ (Darwin 1871). Bis heute werden Variationen bei Lebewesen mehrheitlich als Folge zufälliger Mutationen aufgefasst. Im Folgenden soll jedoch gezeigt werden, dass unsere Intuition auf die richtige Spur führt: Die Erzeugung von neuen Variationen in den Nachkommen von Organismen wird durch Eigenschaften ihres Erbguts ermöglicht, d. h. die Variationen sind im Genom der Eltern angelegt und vererbbar, genauso wie Augen, Ohren, Organe usw.
Die Variation, die Mendel an Erbsen untersuchte, wobei ihm die Entdeckung mehrerer grundlegender Vererbungsgesetze gelang, beruht auf unterschiedlichen Allelen. Eines der sieben Merkmale, die Mendel bei Erbsen beschrieb, nannte er den I-Locus. Dies bezog sich auf die Farbe der Samen. In Mendels Jargon stand „I“ für Dominanz (gelb); „i“ für Rezessivität (grün). Die Pflanzen mit „I“ hatten gelbe Samen, Pflanzen ohne „I“ grüne. Mehr als 150 Jahre danach wurde entdeckt, wie Mendels Gelb-Grün-System auf molekularer Ebene funktioniert. Die Farbe wird durch das stay-green-Gen (abgekürzt: STG) bestimmt, das für ein Protein codiert, das an der Wiederaufnahme von grünen Pigmenten während der Alterung beteiligt ist. Das rezessive Merkmal „i“ ist die mutierte Form des STG-Gens; eine inaktive Variante, die keine Pigmente wieder aufnehmen kann, sodass die Samen ihre grüne Farbe beibehalten. Mendel beschrieb hier die Auswirkungen von nicht-essentiellen oder redundanten Genen (Borger 2022). Dominanz bedeutet, dass mindestens ein redundantes (oder nicht-essentielles) Gen funktionsfähig ist; Rezessivität bedeutet, dass beide Kopien der redundanten (oder nicht-essentiellen) Gene außer Betrieb sind. In diesem Fall entsteht die Variation durch den Verlust von Information. Bei Buntbarschen (Cichlidae) und Silberschwertern (Argyroxiphium) können auch noch zusätzliche Mechanismen wirken, die von Evolutionstheoretikern als standingvariation bezeichnet werden und auf bereits im Genom vorhandener genetischer Information beruhen (Borger 2021, Crompton 2022).
Es sind Eigenschaften des Genoms selbst, die Variation, Anpassung und sogar die Entstehung neuer Arten ermöglichen.
Der als „Lehrbuchbeispiel“ für Evolution bekannte Birkenspanner (Bistonbetularia, s. Introbild) besitzt typischerweise eine hell gesprenkelte Färbung. Vor einigen Jahren fanden Forscher aber heraus, dass die seit etwa 1819 beobachtete dunkle Variante durch einen im Genom vorhandenen genetischen Mechanismus entsteht. Die stärkere Pigmentierung wird durch ein Transposon* verursacht, wenn es sich im Carbonaria-Locus* einnistet (Van‘t Hof et al. 2016). Ein Transposon ist ein genetisches Element (DNA-Sequenz), das sich im Genom umlagern und auf diese Weise andere genetische Varianten erzeugen kann. In diesem Fall wird die Variation durch einen Mechanismus verursacht, der die genetische Information anders anordnet und reguliert, also Ausprägungen bereits vorhandener Informationen steuert.
Die gebogene Schnauze der Bullterrier ist in nur wenigen Generationen entstanden (Abb. 1), denn Fotos von ca. 1930 zeigen, dass die Schnauzen dieser Hunde damals noch gerade waren. Dieser Sachverhalt wird durch sich wiederholende DNA-Elemente (sog. tandem-repeats) erklärt, deren Anzahl sich sehr leicht vermehrt oder verringert (Garner & Fondon 2004). Tandem-repeats sind über das gesamte Genom verteilt und befinden sich sowohl außerhalb als auch innerhalb des codierenden Teils eines Gens. Die Studie zeigte, dass die Zahl der vorhandenen Tandem-repeats große Auswirkungen auf die Gestalt von Hunden haben kann. Bullterrier aus dem Jahr 1930 hatten ein DNA-Element mehr im Runx-2-Gen als ihre heutigen Nachkommen mit gebogenen Schnauzen.
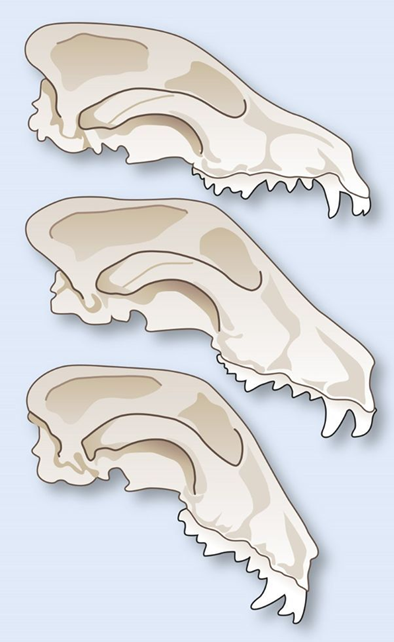
Abb. 1 In nur wenigen Generationen wurde das gerade Gesicht des Bullterriers gebogen. Diese Veränderung der Schädelform wurde von 1930 bis in die 1970er-Jahre durch Züchtung erreicht. In unserem heutigen genetischen Zeitalter wurde entdeckt, dass diese Veränderung mit der Verdopplung eines kleinen genetischen Elements und gleitenden DNA-Polymerasen zusammenhängt (wie im Text erläutert). (Bild: Johannes Weiss nach Garner & Fondon 2004)
Diese und viele andere Beispiele zeigen, dass Anpassungen und Veränderungen sehr schnell erfolgen können, weil keine neuen Gene benötigt werden. Es sind Eigenschaften des Genoms selbst, die Variation, Anpassung und sogar die Entstehung neuer Arten ermöglichen. Wie dies funktioniert, wird erst seit Kurzem verstanden.
TE als Variation-Induzierende Genetische Elemente (VIGE)
Es gibt Mechanismen im Genom, die es den Organismen ermöglichen, sich sehr schnell anzupassen und zu verändern. Die Nobelpreisträgerin (1984) Barbara McClintock war die erste, die darauf hinwies, dass springende genetische Elemente in Mais vorkommen und dort bei Einfügen an eine andere Position in der DNA neue Phänotypen* zur Ausprägung bringen können (McClintock 1950). Sie machte Anspielungen, dass Transposons als genetische Schalter fungieren könnten. Die Genome aller Organismen weisen tatsächlich springende genetische Elemente (Transposons) und andere regulatorische Sequenzen auf (Abb. 2), die lange als Junk-DNA, d. h. als Überbleibsel des evolutionären Prozesses und/oder Invasionen von Viren interpretiert wurden (Orgel & Crick 1980). Um die komplexen Mechanismen zur Erzeugung von Variation durch Transposons zu verstehen, fangen wir mit den Prokaryonten* (Bakterien) an.
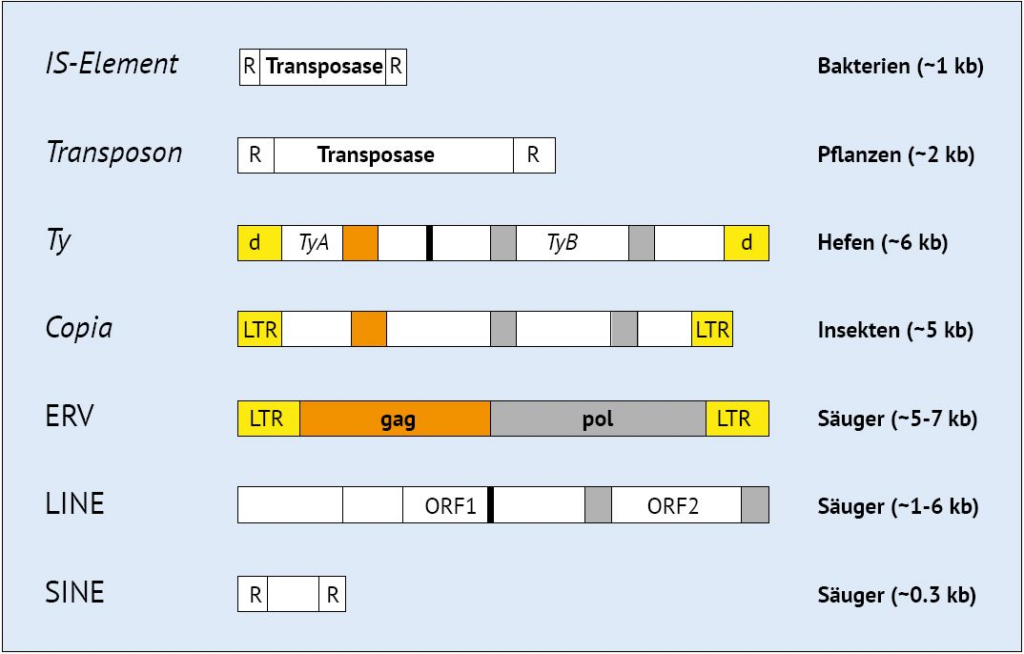
Abb. 2 Unterschiedliche Arten von Variation induzierenden genetischen Elementen (VIGEs) finden sich im ganzen System der Lebewesen, von Bakterien bis zu Säugetieren. In Hefen, Insekten und Säugetieren beobachten wir ähnliche Designs, die jedoch unabhängige Ursprünge haben. Homologe Sequenzen sind durch dieselbe Farbe gekennzeichnet.
In bakteriellen Genomen findet man die sogenannten Insertion-Sequence-(IS)-Elemente, DNA-Transposons, die sich von einer Stelle im Genom zu einer anderen verlagern können. Bakterien besitzen ein Gen, das für das Enzym Transposase codiert, welches die Transposition bewirkt. Dies geschieht in erster Linie durch einen „Cut-Paste“-Mechanismus*, der zu Änderungen in der Struktur und dem Zusammenhang des Genoms führt. Das bekannte Langzeit-Evolutionsexperiment mit dem Darmbakterium Escherichia coli brachte u. a. den Befund, dass veränderte Phänotypen* hauptsächlich auf die Verdopplung und Transposition (Umlagerung) von IS-Elementen zurückzuführen sind (Papadopoulos et al. 1999). Mittels IS-Elementen wurden Gene ein- und ausgeschaltet, regulatorische Sequenzen neu verschaltet oder an andere Stellen eingefügt, was die beobachtete Variation bewirkte. Die Aktivität der IS-Elemente ermöglicht die Überwindung von Stresssituationen, indem sie kryptische (inaktive) Operons* aktivieren, die es dem Organismus ermöglichen, auf eine andere Nahrungsquelle auszuweichen. IS-Elemente bewirken dies auf eine regulierte Weise, indem sie in hungernden Zellen häufiger transponieren als in wachsenden Zellen. In mindestens einem Fall aktivierten IS-Elemente ein kryptisches Operon nur dann, wenn das Substrat für dieses Operon in der Umgebung vorhanden war (Hall 1999). Offenbar erzeugen IS-Elemente gezielt eine für den Organismus nützliche Variation. Sie stellen einen Mechanismus zur gezielten Erzeugung von Variation dar. IS-Elemente sind Variation induzierende genetische Elemente (kurz: VIGE).
In den komplexeren Eukaryonten* sind ebenfalls mehrere Klassen mobiler genetischer Elemente nachgewiesen worden, die als VIGE eingestuft werden können. In der Literatur werden sie heute als transponierbare und transponierte Elemente (kurz: TE*) bezeichnet. Es gibt einfache DNA-Transposons, die den IS-Elementen von Bakterien ähneln, jedoch auch komplexere Retrotransposons* wie die longinterspersednuclearelements (LINE*) und die endogenen Retroviren (ERV*). Sie verfügen über mehr Gene, die sie für ihren Copy-Paste-Mechanismus der genomischen Transposition benötigen. In den Hefen sind die bekanntesten Transposons die Ty-Elemente, in den Insekten sind es die Copia-, Gypsy- und P-Elemente. Und in den Pflanzen beobachtet man die Cin- und Mu-Elemente. Tatsache ist, dass alle Genome unzählige Kopien von TE (VIGE) enthalten. Diese Elemente wurden wie die Transposons lange Zeit als Junk-DNA betrachtet.
Alle bekannten eukaryotischen Retroelemente unterscheiden sich stark von ihren prokaryotischen Entsprechungen, was gegen evolutionäre Verwandtschaft spricht.
Der Name „endogenes Retrovirus“ rührt von der Vorstellung her, es handle sich um Überbleibsel früherer Invasionen von Viren. Die Sichtweise, es handele sich um egoistische DNA, wurde in letzter Zeit stark in Frage gestellt. Genetiker betrachten TE zunehmend als einen wichtigen funktionalen und regulatorischen Teil des Genoms, und man hat eine positivere, sogar symbiotische Rolle für mobile genetische Elemente vorgeschlagen (Faulkner et al. 2009; Upton et al. 2011). Obwohl ihre Rolle in biologischen Prozessen nahezu unbegrenzt zu sein scheint, dienen sie auch bei Eukaryoten vorwiegend der genetischen Variation. Alle bekannten eukaryotischen Retroelemente unterscheiden sich stark von ihren prokaryotischen Entsprechungen (Wells & Feschotte 2020), was gegen evolutionäre Verwandtschaft spricht.
TE (VIGE) in Menschen
Mehrere Klassen von TE (oder VIGE) machen mehr als ein Drittel des menschlichen Genoms aus. Sie können in zwei verschiedene Gruppen unterteilt werden, die DNA- und die RNA-Transposons (Abb. 3). Am besten untersucht sind die RNA-Transposons: ERV und LINE, zwei völlig unterschiedliche Klassen von Retrotransposons*. Beide Elemente werden über sehr komplexe, aber ähnliche Wege als Transposons wirksam. ERV und LINE werden zunächst wie echte Gene in RNA-Moleküle umgeschrieben. Aber im Unterschied zu Protein-codierenden Genen verwenden sie dazu einen anderen Typ von RNA-Polymerase (Typ II statt I). Die RNA wird dann aus dem Zellkern in das Zytosol* transportiert, wo sie durch die Wirkung eines Enzyms (Reverse Transkriptase), das sie selbst codiert, in ein doppelsträngiges RNA-DNA-Hybrid umgewandelt wird. Dieses Hybridmolekül gelangt dann zurück in den Zellkern und wird an einer anderen Stelle wieder in das Genom eingefügt. Das Ergebnis dieses Copy-Paste-Mechanismus sind zwei identische Kopien an verschiedenen Stellen im Genom. Wenn dies in den Keimbahnzellen, in Spermien oder Eizellen geschieht, wird es zu einer stabil vererbbaren Variation. Im menschlichen Genom wurden mehr als 300.000 Sequenzen gefunden, die als ERVs klassifiziert werden können, etwa 8 Prozent der gesamten menschlichen DNA. Ungefähr 500.000 LINE machen weitere 17 Prozent aus (Belshaw et al. 2004).

Abb. 3 Klassifizierung von Transposons. Transposons werden in DNA-Transposons (links) und RNA-Transposons (rechts) unterteilt. Die letztere Gruppe wird wiederum nach ihrer genetischen Struktur unterteilt, d. h. danach, ob sie ein Long Terminal Repeat (LTR) besitzen oder nicht. Die verschiedenen Arten von Transposons, die im menschlichen Genom vorkommen, ihre prozentualen Anteile und ihre geschätzte Anzahl sind angegeben.
Short interspersedelements (SINE) bilden eine weitere Klasse von TE (VIGE). SINE sind durch RNA-Polymerase III (Pol III) transkribierte (übersetzte) Retrotransposons, die nicht über eine eigene Reverse Transkriptase verfügen, sondern nur durch die enzymatische Aktivität von LINE mobilisiert werden können. Sie sind in der Regel 85–500 Nukleotide lang. Eine Million SINEs machen weitere 11 Prozent des menschlichen Genoms aus. Die beim Menschen am häufigsten vorkommenden SINE sind die so genannten Alu-Elemente, sich wiederholende Einheiten von nur drei Nukleotiden* (Elbarbary et al. 2016).
Schließlich gibt es noch die oben erwähnten Tandem-repeats (TR). Es handelt sich um kurze repetitive (sich wiederholende) Sequenzen, die nicht aktiv transponieren können und in der Regel fester Bestandteil der codierenden Region von Proteinen sind – oft ein DNA-Abschnitt, der für Glycin (GGC), Prolin (CCG) oder Alanin (GCC) codiert. Die TR können durch „gleitende DNA-Polymerasen“ während der DNA-Replikation leicht verkürzt oder verlängert werden. Das heißt, die DNA-Polymerasen rücken ein Triplett vor oder zurück. Die bereits erwähnte gebogene Schnauze des Bullterriers ist ein Beispiel für Veränderungen, die von TR hervorgerufen werden können.
TE (VIGE) als genetische Kontroll-Elemente (Promotoren, Enhancer)
Im menschlichen Genom ist derzeit nur die mobile Aktivität der LINE-1 (und der damit verbundenen SINE) nachgewiesen. Sie vervielfältigen sich während der Zellteilung und produzieren dann 100–1000 neue Kopien. Dennoch fungieren nachweislich Tausende ERV, LINE und SINE als regulatorische Elemente der Genexpression, da sie reich an Transkriptionsfaktor-Bindungsstellen* sind. Faulkner et al. (2009) haben nachgewiesen, dass sie als alternative Promotoren* und Enhancer* fungieren: 30 Prozent der RNA-Transkripte von Menschen beginnen innerhalb repetitiver Elemente (Faulkner 2009). Enhancer befinden sich auf dem DNA-Strang vor oder nach dem codierenden Teil des Gens, das sie kontrollieren, meistens weit entfernt davon (nicht selten mehrere 100.000 bp* vom Gen entfernt). Durch die räumliche Anordnung der DNA im Chromatin* kommen die Enhancer–Sequenzen der codierenden Sequenz dennoch sehr nahe und bieten die richtigen Bindungsstellen für die bereits erwähnten Transkriptionsfaktoren*, die bei der Aktivierung der Transkription des Gens helfen. Eine revolutionäre Studie, publiziert im Jahr 2016, legt nahe, dass solche Wechselwirkungen zwischen entfernten Regionen bewirken, dass verschiedene Gene zusammen in kooperierenden genetischen Netzwerken exprimiert werden können (Corradin et al. 2016). Möglicherweise werden diese höheren dreidimensionalen genomischen Strukturen, die Falten und Überkreuzungen, von repetitiven DNA-Sequenzen gebildet (Isochoren). Auch die SINE und ERV unterstützen die Expression von Genen und genetischen Netzwerken. Die oben erwähnten Alu-Elemente finden sich in den Introns*, in den Promotoren und im codierenden Teil von Genen. Auf diese Weise beeinflussen SINE die Expression von Genen. Alu-Elemente sind auch häufig die Auslöser von Rekombinationen und Duplikationen (Lönnig & Saedler 2002; Elbarbary et al. 2016). Die Auswirkungen, die TE (VIGE) auf biologische Prozesse (Genexpression, Rekombination des Genoms usw.) haben, werden oft als Variation eingeordnet.
TE (VIGE) und die Epigenetik
TE haben sich auch als ein Schlüssel zum Verständnis der individuellen Entwicklung (Ontogenese) mehrzelliger Organismen erwiesen. Bekanntlich gehen alle Zellen von Mehrzellern aus einer einzigen befruchteten Eizelle (Zygote) hervor und haben daher alle die gleiche genetische Grundausstattung. Dennoch machen nicht alle Zellen von den gleichen genetischen Programmen Gebrauch. Das ist der Grund für die Unterschiede zwischen Leberzellen, Nierenzellen oder Neuronen. Die Gesamtheit der biologischen Prozesse, die die Zugänglichkeit zu den richtigen Programmen im Genom regeln, wird als Epigenetik bezeichnet. TE spielen eine wichtige Rolle in der Epigenetik, indem sie durch ihre Aktivität und Präsenz in der DNA den Zugang zu den richtigen Programmen steuern. Sie tun dies, indem sie durch „Öffnen oder Schließen“ des Chromatins* Gene mehr oder weniger für die Transkription zugänglich machen (Eu- und Heterochromatin). Auf diese Weise reguliert LINE-1 die globale Zugänglichkeit von Chromatin im frühen Mausembryo unmittelbar nach der Befruchtung (Jachowicz et al. 2017). Dies ist wichtig, weil die normale epigenetische Kontrolle in den ersten 48 Stunden kaum vorhanden ist. Huda & Jordan (2009) beschreiben verschiedene Funktionen von TE in epigenetischen Kontrollprozessen. Sie zeigen, wie TE-Sequenzen Transkriptions-Startstellen in die richtige Position bringen, um die Transkription nahe gelegener Gene einzuleiten. Als Enhancer können sie epigenetische Modifikationen der Histone* bewirken, wodurch das Chromatin für die Transkriptionsmaschinerie besser zugänglich wird. Tatsächlich verfügen stark und breit exprimierte Gene über mehr SINE, während schwach exprimierte Gene häufig von LINEs umgeben sind. Darüber hinaus fungieren TE als sog. Randelemente (engl.: boundaryelements), also als DNA-Sequenzen, die als Puffer zwischen aktiven und inaktiven Chromatinen fungieren. Man vermutet, dass sie damit die Ausbreitung epigenetischer Signale verhindern und die Chromatine im richtigen Zustand halten (Huda & Jordan 2009). Das Genom ist also nicht voller egoistischer DNA, sondern voller Kontrollelemente, deren Aktivität und Position im Genom den genetischen Output und damit auch Variation bestimmen.
Wie Barbara McClintock herausfand, haben Pflanzen viele aktive TE, die sich stark auf den Phänotyp auswirken. Man denke nur an all die Kohlsorten, die gezüchtet wurden (Abb. 4). Pflanzen sind überhaupt hinsichtlich mobiler genetischer Elemente viel besser untersucht. Es gibt eine klare Verbindung zwischen mobilen genetischen Elementen, der lokalen Chromatin-Umgebung und der epigenetischen Regulation der Genexpression. In Pflanzen führt die neue Eingliederung von mobilen Elementen oft zur Bildung von Heterochromatin, einer verdichteten DNA-Struktur, die die Gene unzugänglich macht (und damit inaktiviert). Zum Beispiel haben einige Arabidopsis-Pflanzen (Acker-Schmalwand) ein mobiles Element stromabwärts des BONSAI-Gens, was die Ausbreitung der DNA-Methylierung verursacht, wodurch ein Zwerg-Phänotyp induziert wird. Die durch Transposons vermittelte epigenetische Variation wird durch Umweltveränderungen ausgelöst, wodurch Pflanzen sich schnell an verschiedene geografische Regionen anpassen können.

Abb. 4 Die Genome der Kreuzblütler (Brassicaceae) bestehen zum größten Teil aus transponierbaren und transponierten Elementen. Deren Aktivität hat in hohem Maße zu den verschiedenen Formen der Kohlsorten beigetragen. (Adobe Stock)
Basierend auf der Präsenz, Position und Art von mobilen Elementen gibt es unterschiedliche Varianten von Sojabohnen und Mais, die zu einer frühen oder späten Blüte führen. Sojabohnen und Mais sind ursprünglich tropische Pflanzen, dennoch gedeihen sie auch als Kulturpflanzen in höheren Breitengraden, wo die Tage länger sind. Die Anpassung der Blüten- und Samenbildung an längere Tage geht ebenfalls auf den Einschub eines Transposons zurück, der eine epigenetische Umprogrammierung verursacht. In Mais reguliert ein Transposon stromaufwärts des ZmCCT-Gens die Lichtempfindlichkeit. Der Urform des Mais, der nur in den Tropen gedeihenden Teosinte, fehlt das Transposon an dieser Stelle und sie bildet in der gemäßigten Zone keine Blüten aus. Die Einfügung des Transposons demonstriert, dass mobile Elemente es dem Mais ermöglichten, sich auch in gemäßigten Zonen zu verbreiten. Ein ähnlicher Mechanismus wurde für Sojabohnen beschrieben (Wei & Cao 2016).
Die mobilen Elemente, die die epigenetischen Schalter umstellen und so neue epigenetische Regulierungsmuster erzeugen, sind die Ursache für vererbbare Anpassungen, Varietäten und Raritäten.
Manche Pflanzen besitzen sogar temperatursensitive Transposons. Ein gut dokumentiertes Beispiel ist Onsen. Wird dieses Transposon neben ein bestimmtes Gen eingefügt, wird das Gen erst bei etwas höheren Temperaturen exprimiert. Die Pflanzen, die Onsen im Genom besitzen, können sich schnell an eine sich erwärmende Umgebung anpassen. In einer Zeit, in der die Medien fast täglich den Klimawandel und die mutmaßlichen Herausforderungen für die Organismen thematisieren, zeigt das Onsen-Gen, dass die Pflanzen bereits vorbereitet sind. Mit einem aktiven Onsen-Gen können sie sich in nur wenigen Generationen dauerhaft an die höheren Temperaturen anpassen (Ohama et al. 2017). Anpassung erfolgt hier also nicht sehr langsam gemäß den Vorstellungen des Neodarwinismus. Die mobilen Elemente, die die epigenetischen Schalter umstellen und so neue epigenetische Regulierungsmuster erzeugen, sind die Ursache für viele neue vererbbare Anpassungen, Varietäten und Raritäten.
TE (VIGE) in Lernprozessen
Die Rolle der TE bei der Genregulation ist nahezu unerschöpflich. Überall, wo TE im Detail untersucht werden, entdeckt man neue Funktionen. Eine der wichtigen Funktionen der LINE ist die Steuerung des Aufbaus und der Feinabstimmung der neuronalen Netzwerke im Gehirn (Upton et al. 2015; Corradin et al. 2016; Jönsson et al. 2019). Das Säugetier-Gehirn ist ein äußerst komplexes Organ, das über tausend verschiedene Arten von Neuronen mit einer Vielzahl von Funktionen verfügt. Entgegen der gängigen Lehrmeinung, dass jede Zelle in unserem Körper die gleiche DNA hat, sind die meisten Hippocampuszellen des Gehirns genetisch unterschiedlich. Erst kürzlich fand man heraus, dass die Transposition von LINE-1 die DNA dieser Gehirnzellen gezielt verändert. Auch eine Gruppe von Forschern des renommierten amerikanischen Salk Instituts zeigten, wie LINE-1 Teile der DNA, einschließlich protein-codierender Gene, zielgenau ausschneidet und dadurch Variation in den Neuronen hervorruft. Sie vermuten, dass diese Mechanismen die Expression von Genen beeinflussen, die für das sich entwickelnde Gehirn entscheidend sind (Upton et al. 2015; Corradin et al. 2016; Jönsson et al. 2019).
Transponierbare Elemente fungieren gleichsam als System zur Datenkomprimierung.
Für viele der durch Transposons veränderten Gene wurde bereits nachgewiesen, dass sie bei einer Vielzahl von Verhaltensweisen und Lernprozessen eine Schlüsselrolle spielen. Künstlich herbeigeführte Veränderungen in diesen Genen, die die Wirkung von LINE nachahmen, führten zu Verhaltensänderungen (Treiber & Waddell 2020). Die Erkenntnisse können zu einer Erklärung beitragen, welche Prozesse bei unseren einzigartigen Gedanken und Empfindungen ablaufen und warum auch eineiige Zwillinge so verschieden sein können.
Diese neuen Funktionen von LINE waren völlig unerwartet. Dennoch benötigt das Genom einen Mechanismus, um die enorme Variation von Neuronen zu bewirken. Denn der Hippocampus besteht aus Milliarden differenzierter Neuronen, die sich fast alle voneinander unterscheiden. Das enorme Ausmaß der erforderlichen Variation kann unmöglich in Genomsequenzen codiert werden, da das Erbgut dann viel zu groß wäre. Würden alle Informationen zur Bildung der Millionen unterschiedlichen Neuronen in der DNA-Sequenz im Zellkern gespeichert, müsste dieser völlig überdimensioniert werden, um als Datenverarbeitungssystem funktionieren zu können. TE fungieren sozusagen als System zur Datenkomprimierung.
Gesteuert und überwacht
Ursprünglich ging man davon aus, dass bakterielle IS-Elemente keine oder nur eine geringe Sequenzspezifität aufweisen, was bedeutet, dass der Ort der Integration in der Regel nicht festgelegt ist (von einigen wenigen Ausnahmen abgesehen). Eine Überprüfung der öffentlichen Datenbanken ergab jedoch, dass mehrere IS-Elemente einen subtilen, noch nicht vollständig verstandenen Mechanismus bei der Auswahl des Ziels anwenden (Siguier et al. 2014). Dass es gezielte Mutationen durch Transposons gibt, ist inzwischen unbestritten. Ein interessantes Beispiel ist die Einfügung des IS-5-Transposons an einer bestimmten Stelle stromaufwärts des Glycerin-Operons glpFK in E. coli. Die Insertion aktiviert spezifisch die Expression des GlpFK-Operons, was die Verwendung von Glycerin als Kohlenstoffquelle ermöglicht (Saier & Zhang 2015). Da Bakterien jedoch nur über ein kreisförmiges Chromosom verfügen und sich fast unbegrenzt teilen können, sind eine sequenzabhängige Einfügung und eine strenge Kontrolle über ihre Aktivität möglicherweise nicht erforderlich: In einer Population von Milliarden von Mikroorganismen können alle möglichen chromosomalen Umlagerungen allein aufgrund stochastischer Prozesse auftreten.
Bei Organismen, die sich langsamer vermehren, sind weitere Aspekte zu beachten. Jedes Mal, wenn aufgrund von Umweltveränderungen adaptive Veränderungen erforderlich sind, kann dies nicht dem blinden Zufall und der Selektion überlassen werden. Die Organismen würden an der Herausforderung scheitern, lange bevor die ersten Mutationen selektiert werden könnten. Auch wenn es aufgrund der geringen Datenmenge an Details mangelt, ist klar, dass die TE im Gehirn, die bei Lernprozessen helfen, nicht einfach unkontrolliert umherhüpfen können. Tatsächlich wird ihre Aktivität im Genom selbst reguliert, in der Regel durch DNA-Methylierung und/oder den neulich entdeckten piwi-RNA-pathway* (Wang & Lin 2021). Auch die Tatsache, dass man die Mobilität der LINE beeinflussen kann, zeigt, dass ihre Aktivität sequenzspezifisch ist (An et al. 2006). Heute gibt es für sequenzspezifische Transposons viele Anzeichen. Das R2O1 ist ein LINE, das in einer spezifischen Stelle der 28S ribosomalen DNA bei einer Vielzahl von Tieren gefunden wird. Von Evolutionstheoretikern wird dies als „Beweis“ für eine gemeinsame Abstammung interpretiert. Dass diese Schlussfolgerung nicht zwingend ist, zeigt die Tatsache, dass aus Medeka-Fischen (Oryziaslatipes) isoliertes R2O1 auch spezifisch in die Zielsequenz von Zebrafischen integrierte. Und das nicht nur in Fischen, sondern auch in die gleiche Zielsequenz, die in der DNA des Menschen vorhanden ist (Su et al. 2019). Die Integration ist also nicht zufällig, sondern sequenzspezifisch, und eignet sich somit nicht als Indiz für gemeinsame Abstammung. Es ist inzwischen gut etabliert, dass die LINE-1-Integration im Genom des Menschen bevorzugt an der sogenannten Konsensus-Spaltstelle für das Endonuklease-Enzym erfolgt, die durch einen kurzen Abschnitt von sieben Basenpaaren in der DNA-Sequenz (5‘-TTTTT/AA-3‘) bestimmt wird (Miller et al. 2021). Auch lokale genomische Merkmale wie z. B. bestimmte Chromatinstrukturen sind Hotspots für die Integration von transponierbaren Elementen (Sultana et al. 2017). Die Aktivität des SINE namens Alu-HS6 scheint ebenfalls kontrollierten Mechanismen zu unterliegen, da es im Genom von Mensch, Gorilla und Orang-Utan auf denselben Positionen zu finden ist, nicht aber im Genom von Schimpansen (Abb. 5). Diese unerwartete Beobachtung veranlasste die Forscher, die Möglichkeit in Betracht zu ziehen, dass das Alu-HS6-Element ganz gezielt aus dem Schimpansengenom entfernt wurde (Hedges et al. 1999). Wie bei allen streng kontrollierten genetischen Prozessen sind Dysfunktionen und Krankheiten Folgen von Störungen und Zufallsintegrationen. Eine unkontrollierte Transposonaktivität beeinflusst und fördert tatsächlich den Prozess des Alterns und altersbedingte Krankheiten (Gorbunova et al. 2021). In einem biblischen Rahmen könnte eine entfesselte Aktivität von Transposons also erklären, was nach dem Sündenfall geschah, warum wir altern und schließlich sterben.

Abb. 5 Das genetische Alu-Element HS6 ist beim Menschen, Gorilla und Orang-Utan vorhanden, nicht aber beim Schimpansen und Graukehl-Nachtaffen. Es gibt auch keinen Hinweis darauf, dass an dieser Stelle im Schimpansengenom jemals ein Alu HS6 vorhanden war. Diese Ergebnisse legen nahe, dass die Integration und Exzision (Ausschneiden) dieser genetischen Elemente sehr präzise erfolgt. (Nach Hedges et al. 1999)
Zum Schluss
In der Biologie muss heute bezüglich der Quellen für Variabilität umgedacht werden: Vererbbare Variation ist größtenteils nicht „eine Folge des Zufalls“, sondern auf TE (VIGE) zurückzuführen, die in allen erforschten Genomen reichlich vorhanden sind. Nicht nur Variation, sondern auch Anpassungen und sogar Artbildung gehen demnach maßgeblich auf im Genom vorhandene Elemente und entsprechende (programmierte) Mechanismen zurück (Abb. 6). Aufgrund ihres redundanten Charakters (es gibt Tausende von ihnen) wurden TE oft voreilig als funktionslos abgetan. Es wurde argumentiert, dass es so viele von ihnen gäbe, dass bei Weitem nicht alle eine Funktion haben könnten. Doch sollte bedacht werden, dass die Erzeugung von Variation in den Nachkommen eine Eigenschaft ist, ein funktionales Merkmal, das diffus über das gesamte Genom verteilt ist – und als solches war es sehr schwer zu lokalisieren.
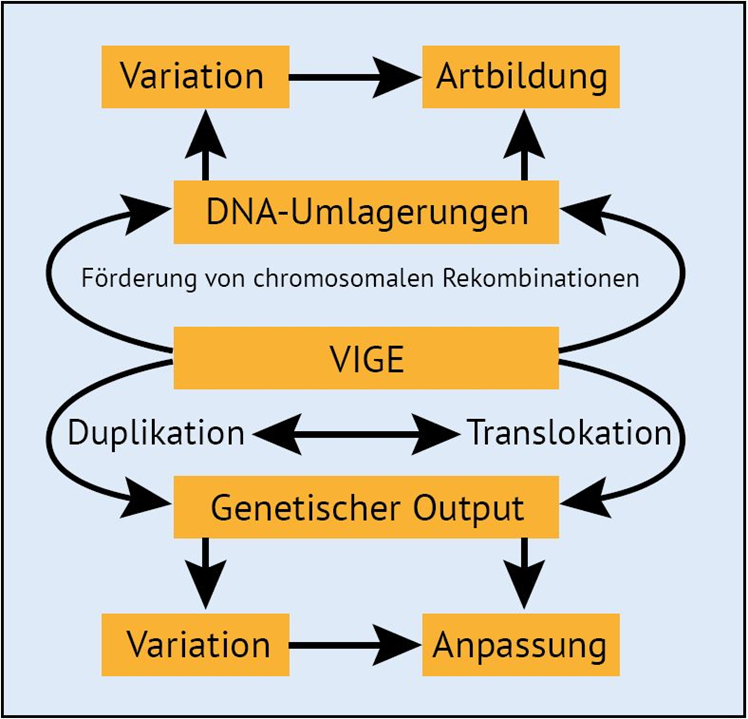
Abb. 6 Schematische Darstellung der zentralen Rolle, die VIGEs bei der Entstehung von Variation, Anpassungen und Artbildung spielen können. VIGEs können aufgrund von Positionseffekten den Output von (morpho-)genetischen Algorithmen direkt modulieren; sie fungieren dann als genetische Schalter. VIGEs, die sich auf verschiedenen Chromosomen befinden, können durch chromosomale Translokationen und andere größere strukturelle DNA-Umlagerungen eine Kreuzungsbarriere erzeugen und so die Artbildung vorantreiben.
Heute können wir aus den beschriebenen Befunden schlussfolgern: Die Geschöpfe wurden mit einer Genetik ausgestattet, die die Variation von innen heraus selbst generiert. Diese Erkenntnis hat weitreichende Auswirkungen auf die Darwin’sche Evolutionslehre: Es gibt keine relevante externe natürliche Ursache für den hypothetischen Evolutionsprozess einer Höherentwicklung. Darwin lag diesbezüglich falsch. Variation, Adaptionen und sogar Arten entstehen in einem Prozess, der nicht durch neue genetische Information, sondern eine gesteuerte Aktivität von Transposons verursacht wird.
Glossar
- bp: Basenpaare.
- Carbonaria-Element: Transposon, das sich seit etwa 1819 in dem Carbonaria-Locus des Birkenspanners befindet.
- Chromatin: Chromatin ist das Material, aus dem Chromosomen bestehen. Es ist ein Komplex aus DNA und speziellen Proteinen, meist Histonen.
- „Copy-Paste“-Mechanismus: Transpositionsmechanismus, bei dem das Transposon über eine RNA kopiert und an anderer Stelle im Genom eingefügt wird. Dies führt zu einem Zuwachs an DNA.
- „Cut-Paste“-Mechanismus: Mechanismus der Transposition, bei dem das Transposon von der DNA ausgeschnitten und an anderer Stelle eingefügt wird. Kein Nettogewinn an DNA.
- Enhancer: DNA-Sequenzen, die mit Promotoren vergleichbar sind, aber im Gegensatz zu diesen nicht unmittelbar stromaufwärts der proteinkodierenden Sequenz liegen, sondern in einem gewissen Abstand.
- Eukaryont (Plural: -en): Organismus mit echtem Zellkern (und mit Zellorganellen).
- Expression (Gene): Ausprägen/Verwirklichen eines Gens im Erscheinungsbild (Phänotyp) eines Lebewesens.
- exprimieren: Ablesen und Umsetzung (Nutzen) von Genen.
- Fitness: „Angepasstheit“. Häufig benutzte Synonyme sind Anpassungs- bzw. Adaptationswert, relative Überlebensrate oder Eignung.
- Genom: Das gesamte Erbgut eines Individuums.
- Histone: Proteine, die die Verpackung der DNA bilden.
- Intron: Nicht codierender Abschnitt der DNA innerhalb eines Gens.
- IS-Element: kurze DNA-Abschnitte (typischerweise in Mikroorganismen), die im Genom mobil sind und sich in die DNA einfügen können.
- Locus (Plural: Loci): In der Genetik die physische Position eines Gens im Genom.
- Nukleotide: Baustein der DNA (DNA-Buchstabe).
- Operon: eine Reihe von genetischen Anweisungen („Programm“) in Bakterien für beispielsweise die Nahrungsverdauung.
- Phänotyp: das Erscheinungsbild, das sämtliche Merkmale eines Organismus umfasst.
- piwi-RNA-pathway: Netzwerk aus RNA-bindenden PIWI-Proteinen und kleinen nichtcodierenden RNAs, den so genannten PIWI-interacting RNAs (piRNAs), die zusammen die Transposon-Aktivität unterdrücken.
- Prokaryont (Plural: -en): (Mikro-)Organismen ohne Zellkern (und ohne Zellorganellen).
- Promoter: eine Nukleotid-Sequenz, die die regulierte Expression eines Gens ermöglicht.
- Retrotransposon: Eine DNA-Sequenz, die sich im Genom über einen Copy-Paste-Mechanismus vermehrt.
- TE (kurz für engl. transposableandtransposedelements): DNA-Sequenzen, die sich unabhängig von der DNA der Wirtszelle in Genomen replizieren können.
- Transkript: Produkt, das nach dem Ablesen der DNA entsteht, ein RNA-Molekül.
- Transkriptionsfaktor: (Komplex von) Proteinen (und manchmal auch RNA), der das Abschreiben (Transkription) eines Gens initiiert, indem er an DNA bindet und einen Ansatzpunkt für die RNA-Polymerase bildet.
- Transkriptionsfaktor-Bindungsstelle: Sequenz in einem DNA-Abschnitt (oder in einem Gen) für die Andockung eines Transkriptionsfaktors, damit die Transkription vorbereitet oder initiiert wird.
- Transposon (Plural: Transposons, oder: Transposonen): Genetisches Element (DNA-Sequenz), das sich im Genom umlagern und somit neue genetische Zusammenhänge erzeugen kann.
- Zytosol: flüssiger Bestandteil des Zellplasmas.
Literatur
An W, Han JS, Wheelan SJ et al. (2006) Active retrotransposition by a synthetic L1 element in mice. Proc. Natl. Acad. Sci. 103, 18662–18667.
Belshaw R, Pereira V, Katzourakis A et al. (2004) Long-term reinfection of the human genome by endogenous retroviruses. Proc. Natl. Acad. Sci. 101, 4894–4899.
Borger P (2021) Standing variation: Das Rätsel der Buntbarsche gelöst? Stud. Integr. J. 28, 63.
Borger P (2022) Über den Entwurf des Lebens: Genetische Redundanz. Stud. Integr. J. 29, 90–99.
Corradin O, Cohen AJ, Luppino JM et al. (2016) Modeling disease risk through analysis of physical interactions between genetic variants within chromatin regulatory circuitry. Nat. Genet. 48, 1313–1320.
Crompton N (2022) Die Radiation der Silberschwert-Gruppe. Indizien für präexistente genetische Programme. Stud. Integr. J. 29, 80–89.
Darwin C (1859) The Origin of Species by means of Natural Selection or The Preservation of Favoured Races in the Struggle for Life. First published by John Murray, London.
Darwin C (1871) The Descent of Man. First published by John Murray, London.
Elbarbary RA, Lucas BA & Maquat LE (2016) Retrotransposons as regulators of gene expression. Science 351, aac7247.
Faulkner GJ & Carninci P (2009) Altruistic functions for selfish DNA. Cell Cycle 8, 2895–2900.
Faulkner GJ, Kimura Y, Daubet CO et al. (2009) The regulated retrotransposon transcriptome of mammalian cells. Nature Genetics 41, 563–571.
Garner JW & Fondon HR (2004) Molecular origins of rapid and continuous morphological evolution. Proc. Natl. Acad. Sci. 101, 18058–18063.
Gorbunova V, Seluanov A, Mita P et al. (2021) The role of retrotransposable elements in aging and age associated diseases. Nature 596, 43–53.
Hall BG (1999) Transposable elements as activators of cryptic genes in E. coli. Genetica 107, 181–187.
Hedges DJ, Callinan PA, Cordaux R et al. (1999) Differential Alu mobilization and polymorphism among the human and chimpanzee lineages. Genome Res. 14, 1068–1075.
Huda H & Jordan IK (2009) Epigenetic regulation of mammalian genomes by transposable elements. Ann. NY Acad. Sci. 1178, 276–284.
Jachowicz JW, Bing X, Pontabry J et al. (2017) LINE-1 activation after fertilization regulates global chromatin accessibility in the early mouse embryo. Nat. Genet. 49, 1502–1510.
Jönsson ME, Brattås PL, Gustafsson C et al. (2019) Activation of neuronal genes via LINE-1 elements upon global DNA demethylation in human neural progenitors. Nat: Commun: 10.3182.
Lönnig WE & Saedler H (2002) Chromosome rearrangements and transposable elements. Annu. Rev. Genet. 36, 389–410.
McClintock B (1950) The origin and behavior of mutable loci in maize. Proc. Natl. Acad. Sci. 36, 344–355.
Miller I, Totrov M, Korotchkina L et al. (2021) Structural dissection of sequence recognition and catalytic mechanism of human LINE-1 endonuclease. Nucleic Acids Res. 49, 11350–11366.
Ohama N, Sato H, Shinozaki K et al. (2017) Transcriptional Regulatory Network of Plant Heat Stress Response. Trends Plant Sci. 22, 53–65.
Orgel LE, Crick FHC (1980) Selbstsüchtige DNA: Der ultimative Parasit. Nature 284, 604–607.
Papadopoulos D, Schneider D, Meier-Eiss J et al. (1999) Genomic evolution during a 10,000-generation experiment with bacteria. Proc. Natl. Acad. Sci. 96, 3807–3812.
Saier MH Jr & Zhang Z (2015) Control of Transposon-Mediated Directed Mutation by the Escherichia coli Phosphoenolpyruvate: Sugar Phosphotransferase System. J. Mol. Microbiol. Biotechnol. 25, 226–233.
Siguier P, Gourbeyre E & Chandler M (2014) Bacterial insertion sequences: their genomic impact and diversity. FEMS Microbiol Rev 38, 865–891.
Su Y, Nichuguti N, Kuroki-Kami A & Fujiwara H (2019) Sequence-specific retrotransposition of 28S rDNA-specific LINE R2Ol in human cells. RNA 25, 1432–1438.
Sultana T, Zamborlini A, Cristofari G & Lesage P (2017) Integration site selection by retroviruses and transposable elements in eukaryotes. Nat Rev Genet 18, 292–308.
Treiber CD & Waddell S (2020) Transposon expression in the Drosophila brain is driven by neighboring genes and diversifies the neural transcriptome. Genome Res. 30, 1559–1569.
Upton KR, Baillie JK & Faulkner GJ (2011) Is somatic retrotransposition a parasitic or symbiotic phenomenon? Mob. Genet. Elements 1, 279–282.
Upton KR, Gerhardt DJ, Jesuadian JS et al. (2015) Ubiquitous L1 mosaicism in hippocampal neurons. Cell 161, 228–239.
Van’t Hof AE, Campagne P, Rigden DJ et al. (2016) The industrial melanism mutation in British peppered moths is a transposable element. Nature 534, 102–105.
Wang C & Lin H (2021) Roles of piRNAs in transposon and pseudogene regulation of germline mRNAs and lncRNAs. Genome Biology 22:27.
Wei L & Cao X (2016) The effect of transposable elements on phenotypic variation: insights from plants to humans. Sci. China Life Sci. 59, 24–37.
Wells JN & Feschotte C (2020) A Field Guide to Eukaryotic Transposable Elements. Annu. Rev. Genet. 54, 539–561.
Aktuelle Wissenschaftsnachrichten zu dem Themenbereich lesen.
Hier geht es zu häufig gestellten Fragen zu diesem Thema.
Zurück zur Rubrik „Grundtypbiologie“.